2023年5月25日,中国科学院再生生物学重点实验室、广东省干细胞与再生医学重点实验室彭广敦课题组在Nature methods上发表了题为Simultaneous profiling of spatial gene expression and chromatin accessibility during mouse brain development的研究论文。文章揭示了一种名为MISAR-seq的空间多组学技术(Microfluidic indexing-based spatial assay for ATAC and RNA-sequencing)。该技术通过微流控芯片依赖的靶向barcode递送系统,在保留细胞空间位置信息的前提下同时实现了细胞内ATAC和RNA两种组学信息的捕获,并成功将其应用在了胚胎期小鼠脑发育机制的研究中,不仅首次构建出了小鼠脑发育的时空多组学图谱,同时也揭示了启动子及增强子形成的顺式调控元件在脑发育中的具体调控机制。
单细胞多组学技术已经可以实现一次性获取同一细胞内的多个组学信息,但也伴随着其空间位置信息丢失,空间组学技术不仅可以很好地保留细胞原位的空间位置信息,还可以同时捕获细胞内的转录组信息。但是,目前的空间组学技术主要局限于单个组学信息的捕获。如果能够开发出一种在同一组织中同时捕获多个组学信息的空间多组学技术,将其用于捕获人体内的所有细胞,并构建出单细胞分辨率的三维数字化人,将会极大的帮助我们解析人类细胞的时空谱系及相关调控机制,从而更好的服务于人体衰老和疾病的治疗。
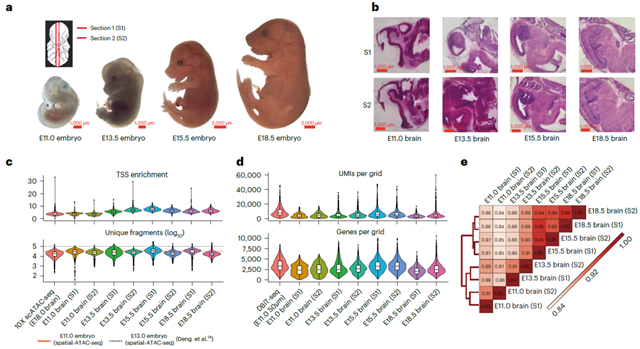
MISAR-seq具有同时在一个空间位置样本上分析RNA与ATAC信息的能力。因此,研究人员特别关注了E11.0、E13.5、E15.5及E18.5的多个胚胎期小鼠脑的发育。首先,研究人员验证了MISAR-seq的数据质量,发现基于空间双组学的ATAC及RNA数据与其相应单组学数据的质量接近甚至更好,每个单元数据点所检测到的基因数大致在2500-3500之间。进一步和单细胞数据的整合分析显示,该技术所捕获到的脑细胞类型与相应单细胞的脑结构注释也非常吻合。
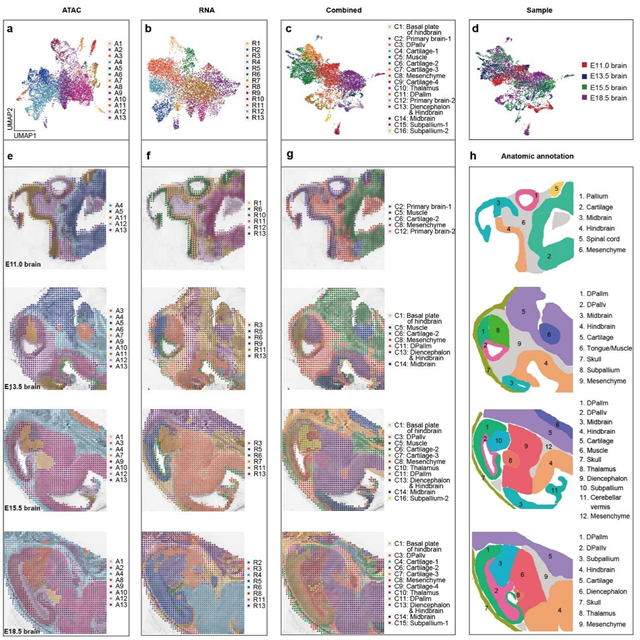
接下来,研究人员将具有不同发育时期且包含两种组学信息小鼠脑发育数据进行了整合分析,并开发了基于空间位置限制与图像特征提取结合的生物信息学工具,有效提高了不同组学在多个不同时期点之间数据的“横向”与 “纵向”整合能力,从而绘制出了小鼠脑发育的时空多组学图谱。结果发现,无论是单独使用MISAR-seq ATAC或RNA数据,或者是同时使用两种组学数据(Combined)进行空间聚类均能较好的进行脑区划分,且聚类结果与Allen Brain Atlas的注释均较为一致。
最后,基于已获得的MISAR-seq数据,研究人员通过绘制两种组学在皮层发育的空间和时间的轨迹,尝试进一步解释皮层发生过程中染色质开放与基因表达之间的级联调控关系。研究人员发现Pax6-Eomes-Tbr1形成的直接级联调控建立了小鼠皮层发生过程中基因表达的顺序和区域特异性。这些结果表明,多组学的时空轨迹分析可以较好的预测决定细胞命运的重要驱动元件,并且可以提供TF的具体相互作用模式,从而对诸如皮层等复杂结构的形成施加精确的调控。
下一步,研究人员期待MISAR-seq能在空间分辨率以及三维组织解析上得到进一步的优化和拓展,且能够兼容更多的组学信息,并基于此开发更好的空间多组学基因调控网络分析工具,从而揭示驱动结构复杂组织中细胞身份和功能决定的具体机制。
研究团队主要由来自中国科学院广州生物医药与健康研究院、生物岛实验室、广州实验室的人员组成。博士生江伏青,钱莹莹、朱淼及实习研究员周鑫等人为该论文的共同第一作者,彭广敦研究员为论文的通讯作者。研究工作还得到了广州实验室崔桂忠研究员、昆明理工大学陈凯研究员的帮助。
图1. MISAR-seq的数据质量
图2. E11.0, E13.5, E15.5 和E18.5小鼠脑的空间基因表达、染色质可及性以及结合二者的空间图谱