重点实验室开发的scDIOR软件打通单细胞组学R/Python平台壁垒
1月6日,中国科学院再生生物学重点实验室陈捷凯课题组开发的单细胞数据IO软件“scDIOR”于BMC Bioinformatics杂志在线发表。该软件统一了多种单细胞分析工具的数据结构,使单细胞数据能够在不同平台间快速转换,减少存储和内存消耗,将显著提升单细胞研究的效率。
单细胞测序应用极广,其数据具有样本量大、信息量大、统计和挖掘极为复杂等特点,科学家往往需要不断切换不同软件,不同平台来满足分析需求。然而,单细胞数据在不同平台之间的传输存在技术障碍,导致科学家将大量的时间花费在数据转换上,严重影响研究效率。
为了解决上述问题,陈捷凯课题组开发了软件scDIOR,统一了R和python平台的三种主流的数据结构,即Seurat,SingleCellExperiment和Scanpy。通过scDIOR,单细胞数据以统一的H5格式保存,无论从哪个平台开始,只需两行代码就可实现单细胞数据在不同工具包之间的快速转换,支持转录组和空间组等多种数据类型(可继续迭代增加),最大程度地保留了原始信息。因此,scDIOR可以快速比较一项分析任务在不同工具包的差异;依托H5文件格式的“组”,提供部分数据读取功能,大幅度减少内存消耗和时间消耗;设计了命令行指令,可实现批量数据转化。综上所述,scDIOR可以应用建立一个标准的单细胞数据结构,将不同工具的优势连接起来,帮助科学家更高效地完成单细胞的研究工作。
本研究在陈捷凯研究员和林立惠博士指导下,由生物岛实验室实习研究员冯辉坚完成。软件已经过大量用户使用优化,可在GitHub下载(链接 https://github.com/JiekaiLab/scDIOR),也欢迎提出宝贵意见(可发至feng_huijian@grmh-gdl.cn)。
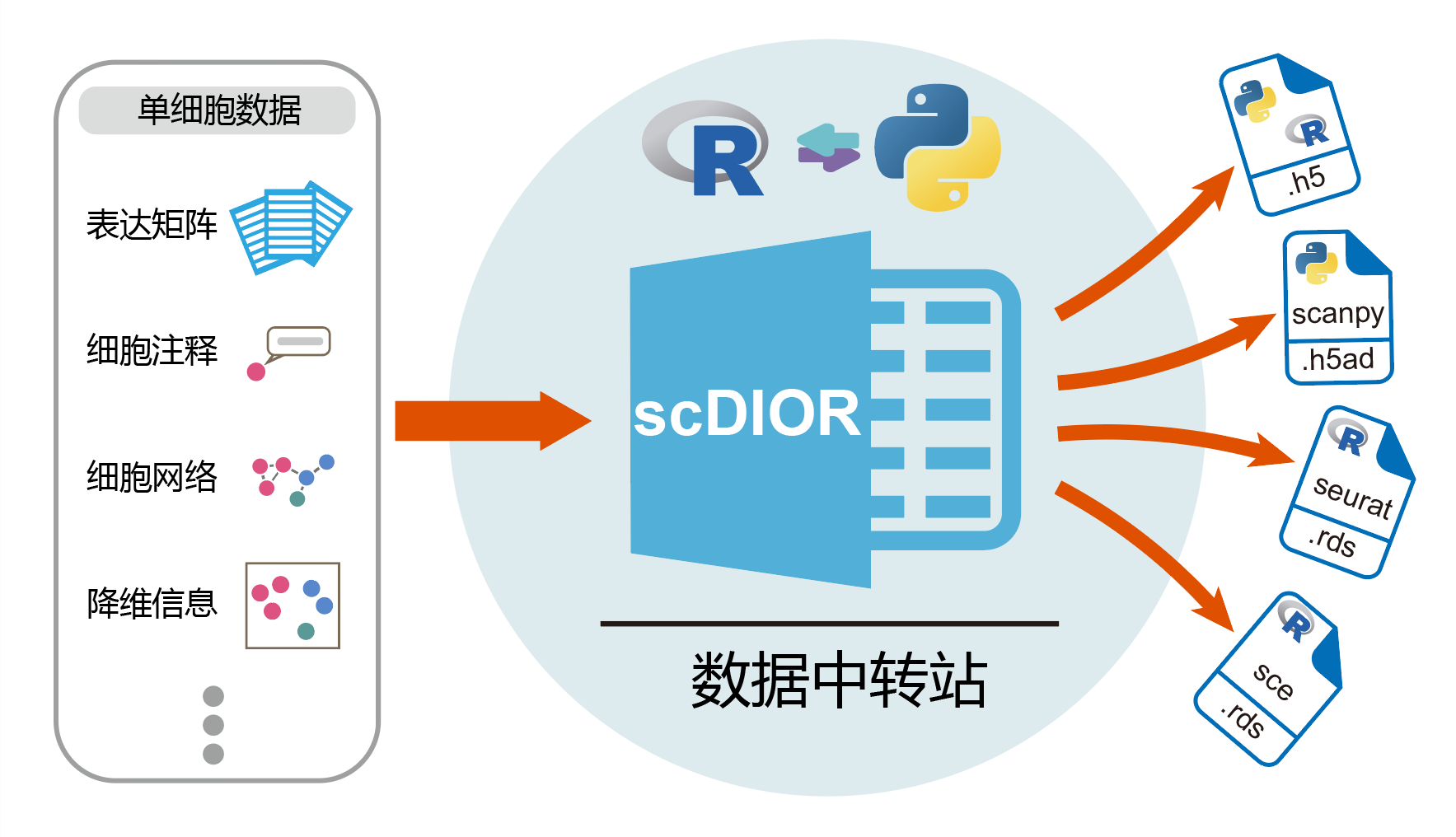
单细胞数据IO软件scDIOR
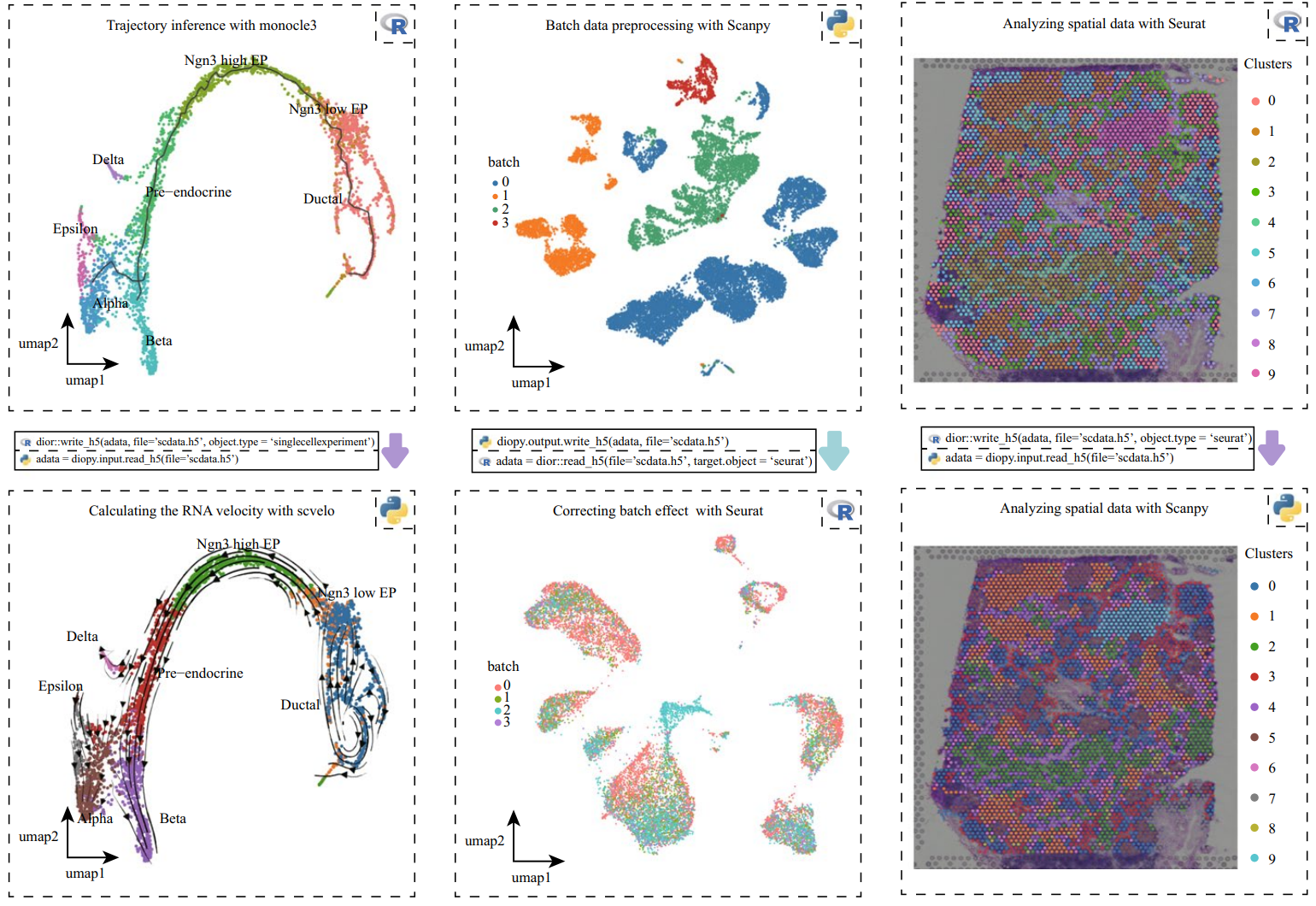
scDIOR很方便地实现数据在平台间的转换,将不同工具的优势连接起来