重点实验室在筛选调控基因表达的“染色质环”新因子取得进展
时间:2020-09-04
来源:中科院再生生物学重点实验室
时间:2020-09-04 来源:中科院再生生物学重点实验室
微米大小的细胞为了储存遗传信息,其遗传物质在细胞核内被紧密地包装和折叠。研究发现基因组的结构是有序的,至少有三个逐级复杂的维度。其中,三维结构是染色质的高级结构,该结构由特殊的结构蛋白质所介导(比如:Cohesin,CTCF等),这些结构蛋白将30nm的染色质纤维折叠成具有“染色质环”的高级结构。“染色质环”不仅有利于精确地保存遗传信息,而且可以介导远距离染色质内和染色质间的相互作用,能将调控元件带到目的基因附近,从而调控基因表达。
9月4日,中科院再生生物学重点实验室、生物岛实验室姚红杰研究员课题组在国际学术期刊Nucleic Acids Research《核酸研究》在线发表了研究成果。该团队通过系统性筛选在基因组上与CTCF共定位的转录因子,鉴定出大量与CTCF存在高共定位率的新转录因子,并选取了转录因子BHLHE40进行后续的功能验证。研究发现BHLHE40可以调控CTCF在基因组上的结合,进而影响其介导的远距离染色质相互作用。
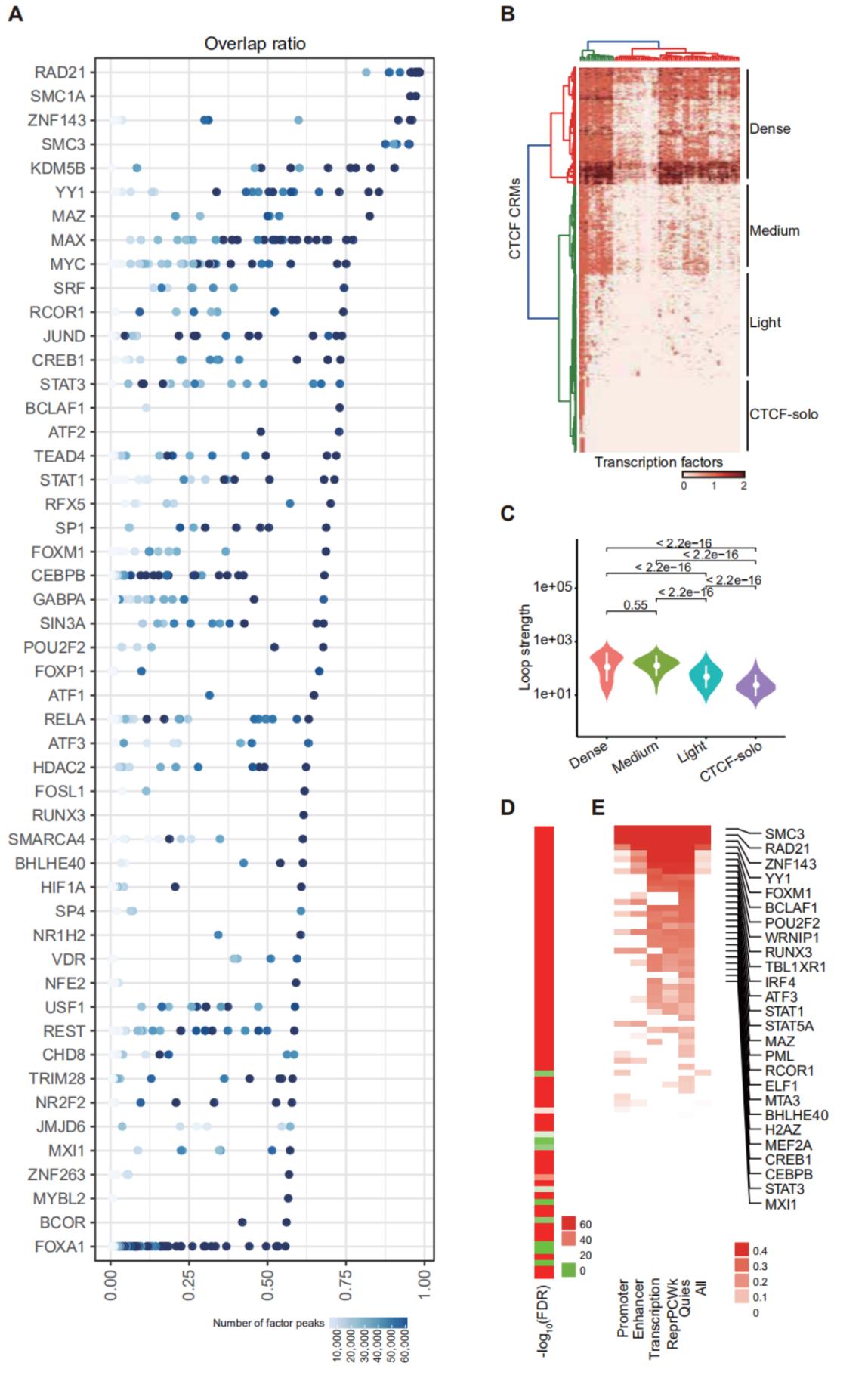
为了鉴定更多调控CTCF结合或CTCF介导的染色质互作的转录因子,研究团队从寻找CTCF共定位因子的角度出发,整合数据库中所有高质量的转录因子ChIP-seq数据,系统性地筛选在基因组上与CTCF具有共定位的转录因子。然而转录因子和CTCF的ChIP-seq数据要在同一细胞、同一处理的前提条件下才具有可比性,但具备这种条件的转录因子ChIP-seq数据非常少。该团队利用CTCF在基因组上的结合具有保守性的特点,通过整合大量细胞和组织中CTCF ChIP-seq数据,首次鉴定出人超保守的CTCF结合位点。这些结合位点在绝大多数细胞和组织中都存在,可以与所有转录因子的ChIP-seq数据进行比较,从而达到系统性筛选CTCF共定位因子的目的。通过这种方法,研究团队鉴定出多种已经报道的与CTCF具有高共定位率的蛋白因子如ZNF143、KDM5B以及Cohesin复合物中亚基RAD21、SMC3和SMC1A等,说明了生物信息分析结果的可靠性。另外还鉴定出了多个之前未见报道的与CTCF具有高定位率的转录因子。
研究团队进一步利用CTCF介导的染色质互作强度信息,与CTCF高共定位率因子的结合强度进行联合分析,发现存在多个共定位因子的CTCF结合位点能够形成更强的染色质互作,暗示存在促进CTCF介导染色质互作的因子。进一步通过相关性分析,鉴定出多个具有调控CTCF介导染色质互作潜能的因子。研究发现敲降BHLHE40显著影响CTCF在基因组上的结合,进而影响CTCF介导的染色质相互作用。
研究团队开发的利用超保守CTCF结合位点鉴定CTCF共定位因子的方法,不仅具有很好的通用性和普适性,而且为后续研究细胞命运决定及疾病发生发展过程中CTCF介导染色质互作的动态变化提供了新思路。
姚红杰研究员为该论文通讯作者,其课题组的胡功成博士、博士生董晓涛和龚士欣为论文共同第一作者。该研究得到南方科技大学Andrew P. Hutchins博士的帮助。该研究得到国家自然科学基金、国家重点研发计划和中国科学院战略性先导科技专项等项目的资助。
系统性筛选CTCF共定位转录因子并鉴定具有调控CTCF介导染色质互作的因子
